
CADERIGE
Présentation du projet
1 Contexte et objectifs
Maintenant que le séquençage n'est plus un point bloquant, les enjeux de la recherche se focalisent sur la compréhension du fonctionnement des génomes et plus spécifiquement sur celui de la modélisation des réseaux d'interactions entre gènes. Dans ce cadre, il s'agit d'établir quelles sont les réactions d'activation ou d'inhibition qui s'établissent entre gènes afin de prédire d'un point de vue qualitatif et quantitatif les caractéristiques des composants produits et donc le fonctionnement de la cellule. Si les travaux effectués en algorithmique, notamment sur le repliement des séquences ou sur la recherche de régularités structurelles, sont capitaux pour établir ces modèles, de tels travaux n'ont cependant de sens que si l'on peut confronter les résultats qu'ils produisent à la lumière des expériences effectuées par les biologistes.
Or, ces résultats sont rapportés, dans leur majeure partie, sous la forme d'articles scientifiques écrits en "langage naturel". Dès lors, l'accès à cette information documentaire est un enjeu central dans la construction des modèles d'interaction entre gènes, car c'est par ce biais que les chercheurs peuvent valider leurs hypothèses, voire définir de nouveaux plans d'expérience. Dans ce cadre, si la recherche d'information à l'aide de mots-clefs offre des performances intéressantes en terme de rapidité de traitement, les résultats renvoyés ne sont pas directement exploitables et nécessitent un important travail d'analyse des documents sélectionnés pour extraire l'information pertinente. L'objectif de CADERIGE est d'automatiser cette extraction en permettant aux biologistes d'exprimer leurs critères d'extraction sous la forme d'une requête dans un sous-ensemble du langage naturel (Figure 1). Le résultat de l'extraction formalisé dans un formulaire pourra être stocké dans une base de données ou de connaissances.
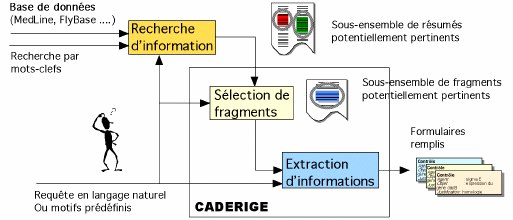
Figure 1. Les trois étapes d'extraction d'information à partir de textes
Par exemple, dans le domaine de la génomique, lorsque qu'un biologiste veut savoir "Quel facteur sigma contrôle l'expression du gène dacB", il pourra obtenir en réponse le fragment de document suivant : "… en ce qui concerne dacB, on observe une bonne ressemblance entre son promoteur et la séquence reconnue par la sous-unité sigma E de l'ARN polymérase …". De manière plus générale, on aimerait pouvoir automatiquement remplir les champs d'une base de connaissances comme ci-dessous :

L'acquisition de ce type de réponse nécessite la mise en œuvre de méthodes d'analyse linguistique et conceptuelle permettant d'interpréter requêtes et documents et de construire dynamiquement les réponses appropriées. Ainsi, dans l'exemple précédent, le système doit prendre en compte une connaissance du domaine exprimant que s'il y a une bonne ressemblance entre le promoteur d'un gène et la séquence reconnue par un facteur Sigma (ce qui est dit dans le fragment de document), alors ce facteur sigma contrôle très probablement l'expression de ce gène (ce qui est demandé dans la requête). Concrètement, le problème d'extraction de connaissances structurées se ramène donc à un problème d'appariement entre la représentation de la requête de l'utilisateur et les représentations des fragments de documents pertinents. Pour ce faire, ces différentes représentations doivent rendre compte les modèles recherchés par les utilisateurs dans les documents, ce qui oblige à effectuer des descriptions au niveau conceptuel. Ce type d'analyse complexe nécessite l'utilisation de thésaurii et d'ontologies spécifiques au domaine d'application. Malheureusement, ces ressources sont, en rège générale, non seulement inexistantes, mais également fort longues à acquérir "à la main".
Du point de vue informatique, le point central du projet CADERIGE concerne donc le développement de nouvelles méthodes automatiques et d'assistances à l'acquisition de telles ressources qui permettront de faire correspondre les modèles répertoriés des utilisateurs à leurs multiples manifestations langagières, de manière à obtenir des représentations conceptuelles canoniques des requêtes et des fragments de texte à apparier. Ces différentes techniques informatiques seront validées en analysant les notices bibliographiques de MedLine concernant l'étude de la transcription des gènes chez la bactérie modèle Bacillus subtilis (Stragier et Losick 96, Hecker et al. 96, de Voos et al. 97) ainsi que chez les deux bactéries lactiques "cousines" Lactobacillus bulgaricus et Lactobacillus sakei en cours de séquençage. À terme, ce projet permettra donc une amélioration notable de l'exploitation qui est faite des ressources documentaires dans le domaine de la recherche d'interaction mais pas exclusivement dans celui-ci (Kodratoff, 99). En effet, dans la mesure où les méthodes informatiques que nous nous proposons de développer sont basées, d'une part, sur l'apprentissage automatique de ressources lexicales et conceptuelles et d'autre part, sur des méthodes génériques d'analyse de la langue, elles seront transposables aux autres disciplines présentant le même type de phénomènes linguistiques. C'est typiquement le cas d'un domaine connexe comme la protéomique, mais plus généralement nos méthodes seront exploitables dans l'ensemble du contexte de l'extraction de connaissance à partir de documentations scientifique et technique.
2. Architecture générale
De manière plus technique, l'architecture du projet CADERIGE s'articule autour de deux grands modules (fig. 2). D'une part, le module d'extraction d'information qui extrait les informations des fragments de textes pertinents et les formalise ; en d'autres termes, ce module implémente le processus que nous venons d'illustrer. D'autre part, le module d'acquisition des ressources linguistiques qui permet lui d'apprendre ou de recueillir de manière semi-automatique l'ensemble des connaissances nécessaires à cette extraction. En pratique, le biologiste exploite le module d'extraction lequel fait appel aux connaissances acquises par la phase d'apprentissage. Le module d'acquisition est utilisé en amont chaque fois qu'il est nécessaire de modéliser un domaine ou de remettre à jour ce modèle.
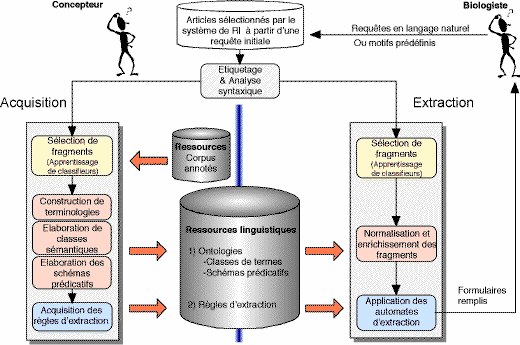
Figure 2. Architecture des modules d'acquisition et d'extraction
2.2 Les résultats
Outre la publication de plusieurs articles scientifiques (Cf. la bibliographie à la fin de cette page), le projet CADERIGE a produit des systèmes informatiques et des jeux de données qui intéressent l'ensemble de la communauté bioinformatique. Le projet s'est officiellement terminé en octobre 2003, cependant les différents partenaires ont continué durant 2004 et maintenant 2005 à développer activement les outils et méthodes mis en place au cours du projet, l'objectif étant de terminer l'implémentation de l'architecture décrite dans la figure 2. Parmi les éléments d'ors et déjà disponibles (n'hésitez pas à nous contacter !) pour la communauté on peut citer :
Classifieurs de texte
Les résultats obtenus sur la sélection des fragments ont été présentés à différentes reprises à des publics de bioinformaticiens. Ils ont reçu un écho très favorable de la part de la communauté. Il s'avère que les classifieurs de textes qui effectuent le tri entre les fragments pertinentes et les fragments non-pertinents peuvent être utilisés indépendamment de la phase d'extraction. La robustesse de cette approche a été éprouvée par des tests sur plusieurs corpus de nature différente et pour des utilisateurs différents. La mise à disposition pour une utilisation à distance (via un site internet spécialisé) est envisagée. Les biologistes auront ainsi à leur disposition des outils pour retrouver automatiquement les fragments de phrases décrivant des interactions.
Editeur XML d'annotation : CADIXE
Au cours du projet est apparue le besoin de développer un outil d'annotation XML pour construire les corpus. Contrairement à ce qui est couramment proposé dans ce type d’éditeur, l’utilisateur n’a pas à structurer directement le texte sous la forme d’un arbre XML. Il peut introduire les balises une à une dans l’ordre où il le souhaite en « surlignant » la partie du texte qui l’intéresse et en y appliquant la balise de son choix parmi celles disponibles. On reste ainsi dans une optique voisine de celle d’un « traitement de texte ». Les zones de textes balisés sont visualisées à l’aide d’une combinaison de polices et couleurs qui sont décrites dans des « feuilles de styles » librement configurables par l’utilisateur. Une description plus complète de l'éditeur est disponible dans les pages "logiciels" de ce site.
Des corpus annotés
Différents corpus sont disponibles ... On peut citer notamment un corpus de 932 phrases identifiées comme pertinentes ou non pour la question des interactions géniques qui est mis à la disposition de la communauté. Ce type de ressource est en effet précieux pour mettre au point les méthodes de traitement automatique (apprentissage ou autres). C'est le corpus pour lequel les meilleurs résultats de classification ont été obtenus et il semble que cela tienne à la rigueur avec laquelle il a été construit. On peut donc faire l'hypothèse que ce corpus pourra servir de référence et de test pour d'autres travaux sur la sélection de fragments et éventuellement pour d'autres tâches.
3 Bibliographie
Publications dans des conférences
BESSIERES P., NAZARENKO A., NEDELLEC C. (2001).Apport de l'apprentissage à l'extraction d'information : le problème de l'identification d'interactions géniques. A paraitre dans les actes de CIDE 2001, 4e Colloque International sur le Document Electronique. Toulouse 24-26 oct. 2001, France. (PFD)
BISSON G., NEDELLEC C., CAÑAMERO L. (2000). Designing clustering methods for ontology building: The Mo'K workbench. Ontology Learning workshop (ECAI 2000), Berlin, 22 août 2000.
BISSON G. ET NEDELLEC C. (2001). Aide à la conception de méthodes de classification pour la construction d'ontologies : l'atelier Mo'K. In H. Brian Eds. Actes des Journées Francophones d'Extraction et de Gestion des Connaissances (EGC 2001), Hermès (Pub.) Nantes. (PDF)
NEDELLEC C., OULD ABDEL VETAH M., BESSIERES P., BRUN C., JACQ J. Reconnaître les fragments de phrases pertinents pour l'extraction d'information dans les textes de génomique, un problème de classification. Actes de la Conférence francophone d'Apprentissage (CAP 2001), PUG (eds). (PDF)
NEDELLEC C. ET NAZARENKO A. (2001). Application de l'apprentissage à la recherche et à l'extraction d'information - Un exemple, le projet Caderige : identification d'interactions géniques. In Actes de la Journée thématique Exploration de données issues d'Internet organisée le 2 mars 2001 au LIPN. Bennani Y., et al. (Eds). (PDF)
NEDELLEC C. ET OULD ABDEL VETAH M. (2001). Modélisation des interactions géniques à partir de textes. Journée Post-Génomique de la Doua (JPGD), Lyon.
NEDELLEC C. ET OULD ABDEL VETAH M., BESSIERES P. (2001). Sentence Filtering for Information Extraction in Genomics, a Classification Problem. To appear in proceeding of 5th European Conference on Principles and Practice of Knowledge Discovery in Databases (PKDD 01). September 3-7, 2001, Freiburg, Germany (PDF)
Présentations du projet
Présentation du projet CADERIGE aux journées organisées les 26 et 27 avril 2001 par XRCE et l'INRIA Rhône-Alpes sur le thème : "Les ontologies en biologie moléculaire et l'extraction d'informations à partir de textes". Ces journées s'inscrivaient dans le cadre de l'action IMPG ("Informatique, Mathématique et Physique pour la Génomique"). (PDF des transparents).
Synthèses & Documents de travail
NEDELLEC C. Synthèse bibliographique sur l'application de l'apprentissage à la recherche d'information. (PDF)
Publication de membres associés au projet
POIBEAU T. (2001). Extraction d'information dans les bases de données textuelles en génomique au moyen de transducteurs à nombre fini d'états. Conférence Terminologie et Intelligence Artificielle (TIA 2001).
Autres références citées dans ce document
DAILLE B. (1994). Approche mixte pour l'extraction de terminologie : statistique lexicale et filtres linguistiques. Thèse d'Informatique. Université de Paris VII. Février 1994.
BRILL E. (1992). A simple rule-based part of speech tagger. In Proceedings of the Third Conference on Applied Natural Language Processing, ACL. 31 March - 3 April 1992. Trento, Italy.
PILLET V. (2000). Méthodologie d'extraction automatique d'information à partir de la littérature scientifique en vue d'alimenter un nouveau système d'information. Thèse de l'université de droit, d'économie et des sciences d'Aix-Marseille.
MITCHELL, T.M. (1997). Machine Learning, Mac Graw Hill, 1997.
QUINLAN J.R. (1992). C4.5: Programs for Machine Learning, Morgan Kaufmann.